|
||||||||||||||||||||||||
|
 |
||||
STUDIO DI PROTEINE
MEDIANTE SPECTROSCOPIA NMR
GUIDA
PRATICA
Marcatura Isotopica
In questa pagina sono riportate diverse strategie di marcatura isotopica
usate per studi di proteine mediante spettroscopia NMR. Ad eccezione della marcatura isotopica in sistemi “cell-free”, la marcatura delle proteine è preferenzialmente effettuata mediante espressione delle proteine in batteri. La marcatura isotopica è ottenuta mediante l’uso, nei terreni di coltura, di particolari nutrienti. Nella gran parte dei casi è usato un terreno minimo contenente tutti i sali e gli elementi necessari alla crescita, ad esclusione di fonti di carbonio e azoto non marcate (ad es. amminoacidi, proteine e zuccheri), che invece verranno introdotte nel terreno mediante composti di azoto e carbonio marcati con isotopi stabili. Marcatura 15N Preparazione di Proteine La produzione di proteine marcate con 15N è semplice e poco costosa. La proteina può essere ottenuta mediante espressione in batteri cresciuti in terreno minimo con supplemento di 15NH4Cl e glucosio. Applicazioni Usando la proteina marcata con 15N, è possibile effettuare esperimenti standard di 2D NMR-HSQC. Gli spettri HSQC della proteina consentono di valutare se la proteina ha una struttura tridimensionale stabile e, in base alla qualità dello spettro, è possibile valutare se sia conveniente procedere con più complesse strategie di marcatura isotopica. Inoltre è anche possibile effettuare esperimenti di NMR, quali ad esempio “N-H residual dipolar couplings” (RDCs) ed altri, che consentono di dare informazioni di dinamica della proteina. Se la proteina è di piccole dimensioni (<~ 150 amminoacidi) è possibile assegnare le risonanze dell’15N del backbone effettuando esperimenti 15N-NOESY e 15N-TOCSY. La 15N –proteina, infine, potrà essere utilizzata per effettuare esperimenti di titolazione con ligandi, noti o potenziali, e valutare il grado di stabilità dei complessi. Reference: Marcatura 15N, 13C Preparazione della proteina La marcatura 15N,13C è comunemente nota come “doppia marcatura” della proteina. La proteina è prodotta mediante espressione in batteri fatti crescere in terreno minimo con supplemento di 15NH4Cl e 13C-glucosio. Applicazioni Questo tipo di marcatura è molto comune e permette di assegnare gli atomi 1H, 13C e 15N sia del backbone che delle catene laterali degli amminoacidi utilizzando esperimenti di NMR-3D. Le assegnazioni delle risonanze consentiranno di calcolare la struttura della proteina in soluzione in modo accurato. Referenza: Marcatura 15N, 13C, 2H Preparazione della proteina La marcatura 15N,13C, 2H è comunemente nota come “tripla marcatura” della proteina. La proteina è prodotta mediante espressione in batteri fatti crescere in terreno minimo con supplemento di 15NH4Cl e 13C-glucosio e usando D2O al posto di H2O. Da questo tipo di espressione si potranno ottenere catene laterali degli amminoacidi della proteina con il 70-80% di deuterio, vi sarà una certa quantità di 1H dovuti al glucosio. Più alti livelli di deuterazione della proteina (circa 95%), potranno essere ottenuti usando 13C,2H-glucosio. Tuttavia, un tale tipo di marcatura risulta essere più costosa ed in molti casi, può essere sufficiente la marcatura meno costosa. E’ da considerare che gli H legati all’N dei gruppi NH sono scambiabili con il solvente. Ciò comporta che vi potrà essere uno scambio con 1H durante le fasi di purificazione della proteina che avvengono in soluzione acquosa. Pertanto, molti dei normali esperimenti basati sugli NH potranno essere anche effettuati sulla proteina triplamente-marcata. Applicazioni La deuterazione è principalmente usata per proteine di grandi dimensioni. Tenendo in considerazione il loro più basso “tumbling” molecolare e le proprietà di rilassamento, esse sono più complesse da studiare via NMR e la qualità degli spettri è peggiore; i picchi possono diventare talmente larghi che non è ottenibile alcuno spettro. Grazie alla deuterazione della proteina, con la rimozione di molti 1H (protoni), vi sarà un notevole miglioramento delle proprietà di rilassamento. L’assegnazione del backbone e dei Cb delle proteine triplamente marcate è possibile usando versioni “out-and-back” degli esperimenti di HNCACB e HN(CO)CACB. E’ da notare che i “chemical shift” del carboni Ca e Cb saranno spostati più di metà di un ppm o più dai loro valori nella proteina marcata con 15N,13C, ciò a causa dell’effetto dell’isotopo deuterio. Referenza: Marcatura IVL - Ile, Val e Leu Amminoacidi con gruppi metilici nelle catene laterali Preparazione della proteina Lo schema di produzione di proteine marcate IVL è quello che produce proteine uniformemente marcate 2H,13C,15N, eccetto per le catene laterali degli amminoacidi Ile, Val e Leu che saranno marcate mediante il seguente schema:
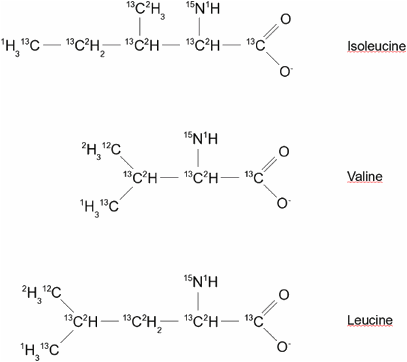
Isoleucina, valina e leucina contengono un
gruppo metilico che è marcato 2H12C e uno che è marcato 1H13C.
Nell’Isoleucina è sempre il gruppo metilico δ1 che è protonato e con 13C,
mentre il gruppo metilico γ2 è deuterato e con 12C. Per la leucina e la valina i gruppi metilici non
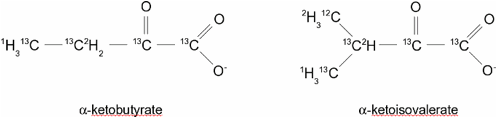
sono marcati stereo-selettivamente.Pagina 3 di 13 Pagina 3 di 13 Questo comporta che metà delle valine /leucine della proteina saranno marcate 13C e 1H ai metili γ1/δ1 e l’altra metà al gruppo γ2/δ2. Per tale motivo, negli spettri NMR, i picchi corrispondenti ai gruppi metilici sia delle valine che delle leucine aumentano, ma solo di circa metà dell’intensità del gruppo metilico d1 della isoleucina. La proteina è prodotta mediante espressione in batteri cresciuti in terreno minimo in D2O usando 13C-2H-glucosio e 15NH4Cl come fonti principali di carbonio ed azoto. Un’ora prima dell’induzione vengono aggiunti α-chetobutirrato e α-cheto-isovalerato (marcati come di seguito riportato) al terreno di crescita per ottenere la marcatura desiderata rispettivamente dei residui di Ile , Val e Leu.
Applicazioni Lo schema di marcatura è stato sviluppato per determinare la struttura di proteine di grandi dimensioni. In genere, delle proteine di grandi dimensioni viene effettuata la tripla marcatura e solo i gruppi NH (principalmente del backbone) saranno 1H- marcati e visibili negli spettri NMR. I NOE saranno così osservabili tra i gruppi NH del “backbone” e ciò comporterà una più bassa qualità delle struttura. Tugarinov e Kay svilupparono lo schema di marcatura IVL, basandosi sul fatto che i gruppi metilici (a) danno segnali forti grazie al “signal averaging”, che (b) sono generalmente presenti nel “core” della proteina; pertanto sono vicini tra loro e sono capaci di dare segnali NOE più forti. Inoltre, tali segnali danno importanti restrizioni strutturali che risultano altamente complementari alle restrizioni NH-NH. Referenza: Marcatura isotopica SAIL - Stereo-Array Preparazione della
proteina La proteina con marcatura SAIL è preparata usando il metodo “cell-free” (cell-free methods) Gli amminoacidi usati per la produzione della proteina sono preparati usando sintesi enzimatiche. La loro marcatura è guidata dai seguenti principi: - in ogni gruppo metilenico, uno degli atomi 1H è streo-selettivamente rimpiazzato da un atomo 2H; - in ogni gruppo metilico, due degli atomi 1H sono rimpiazzati da atomi 2H - i gruppi metilici prochirali di Leu e Val sono stereoselettivamente marcati 12C2H3 e 13C1H2H2 - i sei membri dell’anello aromatico hanno alternativamente metà 12C2H e metà 13C1H. Nel sito SAIL Wiki è possibile osservare la figura illustrante lo schema di marcatura. Applicazioni Il pattern di marcatura SAIL è particolarmente usato per calcolare la struttura di proteine di grandi dimensioni con vantaggi particolarmente visibili negli spettri NOESY. I protoni non scambiabili delle catene laterali degli amminoacidi sono essenziali per definire la loro conformazione, ma danno spesso segnali sovrapposti. Grazie alla riduzione del numero di questi protoni, ottenibile mediante marcatura SAIL dei campioni, la sovrapposizione diminuisce e la qualità degli spettri NOESY aumenta. Inoltre, sebbene vi sia un minor numero di picchi NOE in una proteina ottenuta con marcatura SAIL rispetto a quella uniformemente marcata 15N,13C, è probabile che vi siano picchi NOE di più facile “interpretazione”. In alcuni casi, molti dei picchi che sono rimossi, hanno basso contenuto d’informazioni perché sono a distanza fissa (geminali) o danno informazioni già ottenibili da altri NOE. Inoltre, l’informazione ottenuta è immediatamente stereo-specifica. Un ulteriore vantaggio è che la maggiore fonte di spin diffusione viene rimossa, e pertanto si ha un aumento di accuratezza nella misura delle distanze inter-protoniche. Altri generali vantaggi del campioni SAIL-marcati sono
dovuti al riduzione della larghezza di riga dovuta alla riduzione degli
accoppiamenti “long-range” ed all’eliminazione del “pathway” di rilassamento
dipolare. La strategia della marcatura
degli anelli aromatici rimuove accoppiamenti di legame 13C-13C,
che spesso complicano l’analisi degli spettri o richiedono l’uso di metodi di
analisi di dati di “time constant” per ridurre la complessità
spettrale. Le misure degli accoppiamenti sono fatte più facilmente perché i gruppi
CH2 e CH3 sono convertiti in gruppi CHD e CHD2. La marcatura dei gruppi metilici e metilenici nelle proteine marcate con il metodo SAIL rende anche tali gruppi disponibili per effettuare esperimenti di rilassamento, diretto al fine valutare i moti delle catene laterali.
Referenza: SAIL Wiki dai laboratori di Kainosho, Jee and Güntert Marcatura
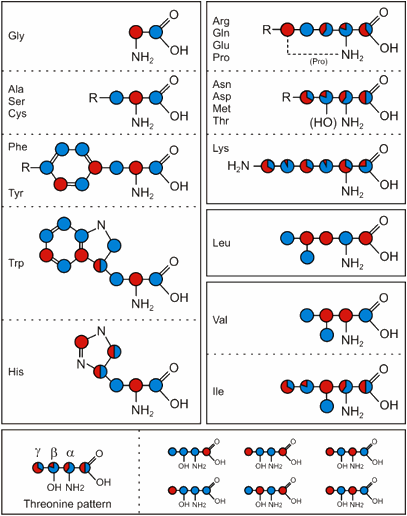
con 1,3-13C- e 2-13C-Glicerolo Preparazione della proteina La proteina è prodotta mediante espressione in batteri cresciuti in terreno minimo con supplemento di 15NH4Cl e o di glicerolo marcato1,3-13C o di glicerolo marcato 2-13C. Lo schema di marcatura ottenuto è un po’ complesso. Nella
figura seguente, i siti colorati in blu sono marcati nel campione marcato con
glicerolo 1,3-13C e i siti colorati in rosso sono marcati nel
campione ottenuto con glicerolo 2-13C.
Per gli amminoacidi sintetizzati via ciclo dell’acido citrico sono ottenibili diversi, così chiamati, isotopomeri, che poi risultano nella marcatura media mostrata sopra. Per la treonina sono mostrati gli isotopomeri individuali che portano un aumento della marcatura media. Una figura completa di tutti gli isotopomeri è ottenibile qui (here). È da notare che le popolazioni relative di isotopomeri, mostrata in queste figure, sono state determinate per la proteina SH3 (Castellani et al. 2002); per altre proteine esse possono essere leggermente diverse. Applicazioni Originariamente la
marcatura con glicerolo 2-13C è stata utilizzata per le misure di rilassamento in soluzione (LeMaster e Kushlan,
1996). Gli atomi Cα, in tali campioni, non
contengono atomi vicini
marcati con 13C (con
la sola eccezione della valina). Misure di rilassamento in gruppi isolati 13Cα-1Hα possono, quindi, essere interpretate più
facilmente, senza dover prendere in considerazione
accoppiamenti degli atomi di carbonio vicinali. Recentemente Castellani et al. (2002) ha mostrato che i campioni marcati con glicerolo 1,3-13C e 2-13C sono utili per la determinazione della struttura delle proteine a stato solido MAS NMR. I campioni hanno diversi vantaggi: (a) molti accoppiamenti-J vengono rimossi e ciò comporta picchi più stretti, (b) accoppiamenti dipolari molto forti tra atomi vicini vengono rimossi e permettono di osservare correlazioni a lungo raggio attraverso deboli accoppiamenti dipolari e (c) la sovrapposizione spettrale viene ridotta. Attualmente questa è, probabilmente, l'applicazione più comune della marcatura 1,3-13C e 13C-2-glicerolo. I caratteristici “pattern” di marcatura per ogni amminoacido possono anche essere usati per effettuare l’assegnazione (Higman et al. 2009). Hong e Jakes (1999) sostituirono
la marcatura con 2-13C-glicerolo con la
marcatura di aminoacidi specifici
(amino
acid specific labelling) al fine di marcare
solo la metà del amminoacidi
con 2-13C-glicerolo. Referenze: Marcatura con Amminoacidi Specifici Preparazione della proteina La proteina è prodotta da espressione in
batteri che vengono
cresciuti in terreno minimo arricchito con piccole quantità di 15NH4Cl e glucosio marcato con 13C, e
con amminoacidi marcati e non. L'idea alla base di tale marcatura è
quella che solo gli
aminoacidi che si
aggiungono in forma marcata
saranno marcati nella
proteina. Sfortunatamente, questo non
sempre porta ad ottenere la marcatura che si desidera, a
causa del metabolismo di E. Coli,
ed il catabolismo provoca un certo grado di interconversione tra amminoacidi. Pertanto, non è possibile creare un
campione proteico con qualsiasi combinazione
di amminoacidi marcati. La situazione può essere
leggermente migliorata utilizzando
ceppi batterici autotrofi od utilizzando inibitori degli enzimi. Pertanto, se si vuole
ottenere un controllo completo dell’incorporazione degli
amminoacidi, devono essere utilizzati
i metodi “cell-free” (cell-free methods). Un modo più economico della marcatura solo di alcuni amminoacidi,
spesso chiamata marcatura inversa, comporta l’espressione
da batteri che sono coltivati in terreno minimo arricchito con 15NH4Cl e glucosio marcato con
13C, nonché amminoacidi privi di marcatura.
Questo elimina la
marcatura di questi amminoacidi
e solo quelli che
non sono stati aggiunti senza marcatura
saranno sintetizzati dai batteri, utilizzando come fonte di carbonio
il glucosio- 13C. Anche in questo caso, può accadere che avvenga una certa quantità di
“scrambling”. Applicazioni La marcatura di specifici amminoacidi
di solito è usata per ridurre la sovrapposizione
spettrale e permettere di monitorare alcuni
amminoacidi senza interferenze dovute ad
altri segnali. In
alcuni casi può anche essere usata per semplificare il processo di
assegnazione. Referenza: Sistema Cell-free Preparazione della proteina Piuttosto
che esprimere la proteina in una cellula, la proteina è espressa in vitro.
Il DNA o l’mRNA della proteina sono immessi in un estratto cellulare contenente
tutto il complesso per la trascrizione e la traduzione ed una varietà di altri
composti, tra cui 20 aminoacidi, nucleosidi trifosfati (NTP), diversi enzimi,
tamponi, sali ecc. La miscela di reazione è in genere di pochi ml-ml, ma i rendimenti che si possono ottenere sono di
vari mg di proteina per ml di miscela di reazione. Chaperoni, detergenti e
altri composti che facilitano il “folding” proteico possono anche essere
aggiunti. Tale marcatura è molto conveniente e semplice
per poter marcare singoli aminoacidi;
basterà, infatti, semplicemente aggiungerli alla miscela di reazione in forma
di amminoacidi marcati ed aggiungere gli altri aminoacidi senza marcatura.
Anche se gli amminoacidi marcati sono piuttosto costosi, i piccoli volumi delle
miscele di reazione usati per l'espressione proteica nel sistema “cell-free” consentono
di non raggiungere costi troppo elevati. Applicazioni L’espressione proteica in sistemi “cell-free” ha diversi
vantaggi, sia per la creazione di specifici programmi di marcatura isotopica, ma anche, in generale, per l'efficienza dell’espressione. La marcatura di uno specifico amminoacido è possibile senza “scrambling” dovuto
al catabolismo in E.
Coli ed all’anabolismo. Inoltre, è
possibile introdurre nelle proteine amminoacidi alternativi, ad esempio fluoro-triptofano, con efficienza molto maggiore rispetto a quando si utilizzano sistemi di
espressione in cellule. Più in generale,
l’espressione proteica in sistemi “cell-free” può essere molto
utile per le proteine che non si esprimono bene nelle cellule, ad
esempio perché sono tossiche per la cellula. Espressione senza cellula si sta rivelando di successo per le proteine di membrana, la cui espressione in grandi
quantità, è noto, può essere
molto difficile da ottenere nelle cellule. Referenze: G. Zubay (1973)
Ann. Rev. Genet. 7 267-287. (Link to Article) T.
Kigawa, Y. Muto and S. Yokoyama (1995) J. Biomol. NMR 6
129-134. (Link to Article) Marcatura di Segmenti Preparazione della proteina Le proteine con marcatura segmentale sono marcate solo
lungo una regione della catena di amminoacidi (in
genere le zone N-o C-terminali). La
preparazione del campione si
suddivide in tre grandi fasi. La Native Chemical Ligation (NTL) prevede la preparazione di
una sezione marcata e una non marcata della catena polipeptidica, utilizzando un peptide di sintesi in fase
solida (SPPS). Queste due sezioni sono
poi unite mediante legame chimico. Questo metodo è limitato a polipeptidi relativamente corti che possono essere sintetizzati
chimicamente con SPPS.
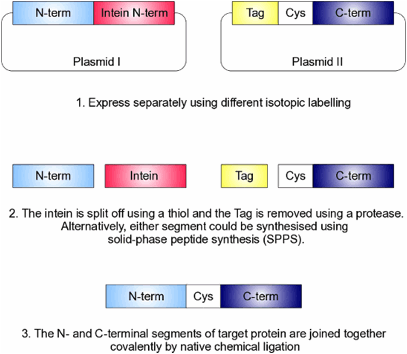
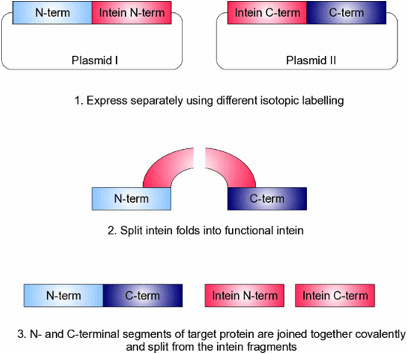
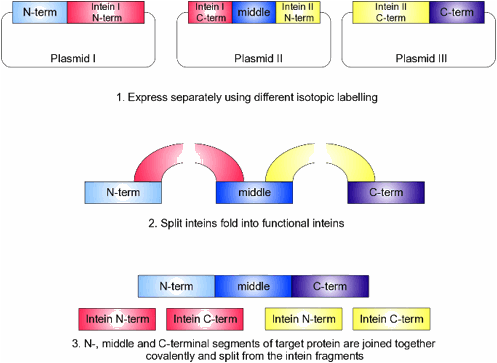
Nel Protein trans-Splicing (PTS), la fase di legame
è ottenuta da uno
spiazzamento dell’inteina funzionalmente ricostituita.
Un’inteina può essere covalentemente legata tra i due segmenti polipeptidici, ed escindere se stessa. Per “trans-splicing” della proteina, le parti N-terminali della proteina e dell’inteina sono
codificate come un’ unica catena polipetidica in un plasmide ed
espresse. I segmenti C-terminali della proteina e dell’inteina
sono, parallelamente, codificati in un secondo plasmide e vengono espressi separatamente, utilizzando
una diversa marcatura isotopica. Quando le due catene proteiche-intein vengono miscelate nelle giuste condizioni, l’inteina può piegarsi nella sua forma funzionalmente corretta e può legare
covalentemente i due segmenti di proteina “target”.
Come con EPL un residuo
di Cys deve essere presente l'unione di due segmenti; perché la reazione di “trans-splicing”avvenga
in modo efficiente, i residui che
fiancheggiano la Cys devono essere accuratamente
scelti in base all’Inteina che viene utilizzata. Se questo richiede mutazioni o inserimenti nella proteina nativa, è consigliabile, verificare che questi non
influenzino significativamente sia la struttura che la funzione della proteina.
Applicazioni Referenze: T. Otomo,
N. Ito, Y. Kyogoku and T. Yamazaki (1999) Biochemistry 38 16040-16044.
(Link to Article) ALTRE MARCATURE Sono qui riportati alcuni altri schemi
di marcatura noti - ma questo non è comunque un
elenco completo. Si prega di
guardare i riferimenti
per ulteriori dettagli. Marcatura dei metili dell’Alanina Un'estensione / alternativa al metodo di marcatura IVL e la marcatura in cui i residui di alanina sono marcati con 13C alla posizione Cβ e deuterati nella posizione Hα. Referenza: Marcatura con 10% di
Glucose Questo si traduce in un sistema di marcatura piuttosto complicato un po 'come la marcatura con glicerolo. Può essere utilizzato per rendere possibile l'assegnazione stereospecifica dei gruppi metilici della Val e della Leu e semplifica anche esperimenti di correlazione 13C-13C MAS-NMR. Referenze: D. Neri,
T. Szyperski, G. Otting, H. Senn and K. Wüthrich (1989) Biochemistry 28
7510-7516. (Link to Article) M. Schubert, T.
Manolikas, M. Rogowski and B.H. Meier (2006) J. Biomol. NMR 35 167-173. (Link to Article) 1-Glucose Labelling Questo schema introduce
atomi 13C isolati
in catene laterali
aromatiche. Questo è stato utilizzato per studiare la dinamica di
catene laterali aromatiche
con metodi di
rilassamento. Referenza: Marcatura con Acid Succinico
Acido succinici marcati [1,2,3,4-13C], [1,4-13C] o [2,3-13C] sono stati utilizzati come fonti di carbonio nel terreno di crescita per i batteri per marcare un complesso di raccolta della luce nei batteri fotosintetici di R. acidophila. Gli spettri di NMR MAS ottenuti mostrano un aumento della risoluzione spettrale ed una ridotta sovrapposizione di picchi. Referenza: Marcatura con Acetato
La proteina è prodotta usando una miscela al 15% di 13C1-acetato, al 15% di 13C2-acetato ed al 70% di 12C1, 12C2-acetato. Questo riduce (ma non elimina) il numero di atomi legati direttamente al 13C e semplifica gli studi di rilassamento. Referenza: |
|||||
| Spectra 2000 Srl Via Santa Margherita di Belice, 16 - 00133 Roma |
E-mail: info@spectra2000.it |